Nucleic Acids Research|郑凌伶与冯枭团队构建大规模跨物种sup-tRNA百科全书ENSURE
核糖核酸(RNA)既承担遗传信息的传递,又参与广泛的调控网络,是连接基因型与表型的关键分子。抑制性转运RNA(suppressor tRNA,sup-tRNA)作为一类特殊的RNA,能够“通读”遗传密码中的终止指令,其在基础研究和应用开发中的潜力巨大,日益受到关注。
近日,我院郑凌伶教授与冯枭副教授团队在国际权威期刊Nucleic Acids Research上发表题为“ENSURE: the encyclopedia of suppressor tRNA with an AI assistant”的研究论文,构建了首个跨物种的抑制性转运RNA百科全书ENSURE,并集成了大型语言模型驱动的AI助手,为突变功能研究、密码子扩展与生物制造机制解析提供重要支持,推动靶向tRNA的基础研究和应用转化。

提前终止密码子(Premature Termination Codons, PTCs)会导致蛋白翻译提前终止,产生截断且失活的蛋白产物。sup-tRNA由于能够识别并“通读”这些异常终止信号,从而在翻译层面恢复全长蛋白质的表达,成为了RNA领域重要的功能元件与工程化对象。随着遗传密码扩展(Genetic Code Expansion)、遗传密码压缩(Genetic Code Contraction)与tRNA生物学的迅速发展,天然与工程化sup-tRNA相关的数据快速积累,亟需系统化整合与标准化注释的公共平台。
为此,研究团队开发了综合性百科全书式平台ENSURE。该平台整合了来自25个物种1200余条分子的多维信息,涵盖疾病与癌症相关突变记录、经实验验证的天然sup-tRNA、工程化tRNA设计与改造策略以及tRNA功能元件与修饰注释等。所有序列均经过多序列比对、tRNAscan-SE二级结构预测与AlphaFold 3三维建模,实现序列—结构—功能的可视化比对与综合分析。ENSURE平台还配备了人工智能AI工具,可为用户提供快速条目检索、术语释义及页面内容导览,显著提升数据浏览与文献对照效率。
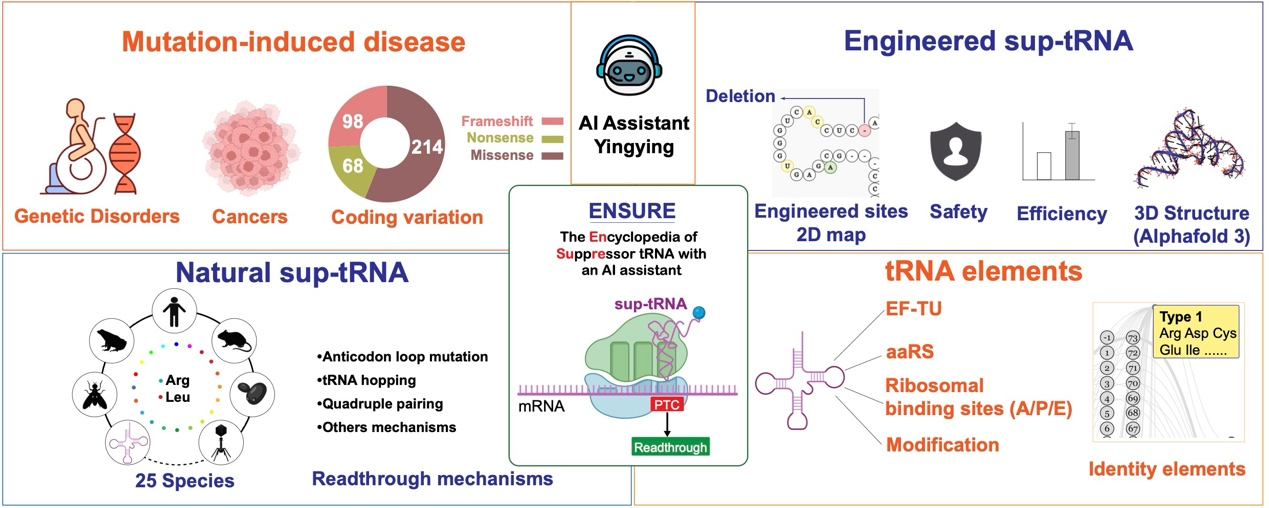
图1 sup-tRNA百科全书ENSURE概览
ENSURE首次将天然与工程化sup-tRNA系统化整合,构建了从序列到结构、从突变到功能的全面关联框架。该平台为sup-tRNA的进化研究、结构设计与工程化改造,以及在生物医药研究、合成生物学与生物制造等领域的技术开发提供了关键数据支撑。团队正在持续更新平台内容,扩展实验验证数据,并完善结构注释与交互式分析功能,促进sup-tRNA 在基础研究与应用技术中的广泛推广与规范化使用。
我院教师郑凌伶教授与冯枭副教授为共同通讯作者,研究生欧阳卓与张怡峰为共同第一作者,本科生曾旭东和吴秋晖等人参与了本研究工作,中山大学李斌副教授、温砚子博士、伦照荣教授、屈良鹄教授提供了重要建议和帮助。研究工作得到了国家重点研发计划、国家自然科学基金和广东省自然科学基金的经费资助。
郑凌伶教授团队长期致力于非编码调控元件挖掘及计算平台开发,建立了全面的非编码RNA数据资源,包括tModBase、deepBase 2.0,dreamBase,ColorCells, tsRFun, exomiR-Explorer等,并开发了专门从高通量测序数据中挖掘RNA及其修饰特征的工具。
ENSURE平台链接:https://trna.lumoxuan.cn/
论文网址链接:https://doi.org/10.1093/nar/gkaf1062
初稿:冯枭 初审:郑凌伶 审核:辛国荣 终审:杨德胜



